题外话¶
Unet是受到FCN启发针对医学图像做语义分割,且可以利用少量的数据学习到一个对边缘提取十分鲁棒的模型,在生物医学图像分割领域有很大作用。
网络架构¶
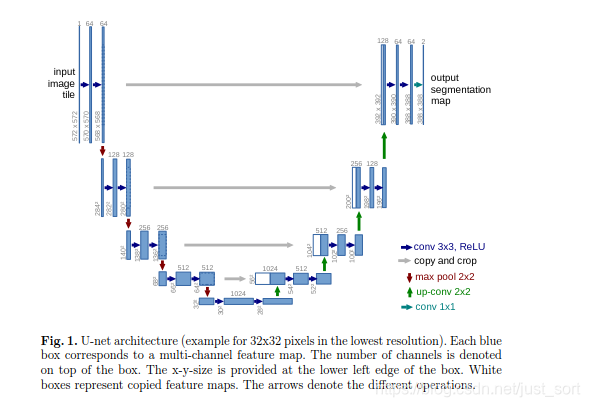
这就是整个网络的结构,大体分为收缩和扩张路径来组成。因为形似一个字母U,得名Unet。收缩路径仍然是利用传统卷积神经网络的卷积池化组件,其中经过一次下采样之后,channels变为原来的2倍。扩张路径由2 * 2的反卷积,反卷机的输出通道为原来通道数的一半,再与原来的feature map(裁剪之后)串联,得到和原来一样多的通道数的feature map,再经过2个尺寸为3 * 3的卷积和ReLU的作用。裁剪特征图是必要的,因为在卷积的过程中会有边界像素的丢失。在最后一层通过卷积核大小为1 * 1的卷积作用得到想要的目标种类。在Unet中一共有23个卷积层。但是这个网络需要谨慎的选择输入图片的尺寸,以保证所有的Max Pooling操作作用于长宽为偶数的feature map。
Trick1: 对于尺寸较大的图像:Overlap-tile strategy¶
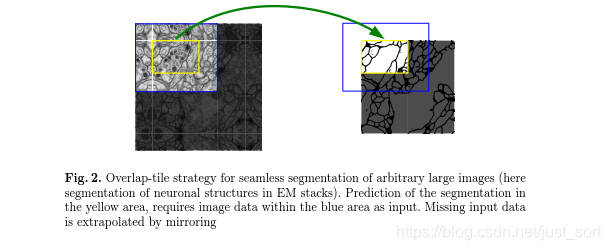
由于网络没有全连接层,并且只使用每个卷积的有效部分,所以只有分割图像完全包含在输入图像中可以获得完整的上下文像素。而这个策略允许通过重叠区块无缝分割任意大的图像,为了预测图像边界区域中的像素,通过镜像的输入图像来外推丢失的上下文。这种平铺策略对于将网络应用于大图像很重要,否则分辨率将受到GPU内存的限制。
数据集可以用数据量较少¶
可用的训练数据非常少,通过对可用的训练图像应用弹性变形来进行数据增强。这允许网络学习到这种变形的不变性,而不需要在注释的图像语料库中看到这些变换。这在生物医学分割中尤其重要,因为变形是组织中最常见的变化,并且可以有效的模仿真实的变形。Dosovitskiy等人已经证明在无监督表征学习的范围内学习不变性的数据增加的价值,通过在一个3*3的粗糙网格中使用一个随机位移向量产生一个平滑的变形,位移量从高斯分布中取样,高斯分布有10个像素的标准差,每个像素的偏移值通过bicubic interpolation来获得。
相同物体间的间隔不容易分割出来¶
很多细胞分割任务中的一大挑战是分离同一类接触体,本文采用加权损失,其中接触单元之间的分离背景标签在损失函数中获得大的权重。以此方法提升对于相互接触的相同物体之间缝隙的分割效果。
主要贡献¶
在医学影像领域,由于数据量本身就很少,这篇论文有效的提升了使用少量数据集进行训练检测的效果,还提出了处理大尺寸图像的有效方法。
总结¶
似乎很多人使用Unet来做边缘检测,这个体现为,我们有什么样的Mask就会获得什么样的边缘。本人已经用Unet训练了自己的数据集,并且得到了预期的结果,之后会提供一个基础,详细的教程来说说如何训练Unet之类的语义分割网络。
代码实现¶
#coding=utf-8
from keras.models import *
from keras.layers import *
def Unet (nClasses, optimizer=None, input_width=512, input_height=512):
input_size = (input_height, input_width, 3)
inputs = Input(input_size)
conv1 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(inputs)
conv1 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv1)
pool1 = MaxPooling2D(pool_size=(2, 2))(conv1)
conv2 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool1)
conv2 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv2)
pool2 = MaxPooling2D(pool_size=(2, 2))(conv2)
conv3 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool2)
conv3 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv3)
pool3 = MaxPooling2D(pool_size=(2, 2))(conv3)
conv4 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool3)
conv4 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv4)
drop4 = Dropout(0.5)(conv4)
pool4 = MaxPooling2D(pool_size=(2, 2))(drop4)
conv5 = Conv2D(1024, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(pool4)
conv5 = Conv2D(1024, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv5)
drop5 = Dropout(0.5)(conv5)
up6 = Conv2D(512, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(drop5))
merge6 = concatenate([drop4,up6], axis = 3)
conv6 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge6)
conv6 = Conv2D(512, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv6)
up7 = Conv2D(256, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv6))
merge7 = concatenate([conv3,up7], axis = 3)
conv7 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge7)
conv7 = Conv2D(256, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv7)
up8 = Conv2D(128, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv7))
merge8 = concatenate([conv2,up8], axis = 3)
conv8 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge8)
conv8 = Conv2D(128, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv8)
up9 = Conv2D(64, 2, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(UpSampling2D(size = (2,2))(conv8))
merge9 = concatenate([conv1,up9], axis = 3)
conv9 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(merge9)
conv9 = Conv2D(64, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv9)
conv9 = Conv2D(nClasses, 3, activation = 'relu', padding = 'same', kernel_initializer = 'he_normal')(conv9)
o_shape = Model(inputs, conv9).output_shape
outputHeight = o_shape[1]
outputWidth = o_shape[2]
conv9 = Reshape((nClasses, input_height * input_width))(conv9)
conv9 = Permute((2, 1))(conv9)
conv10 = (Activation('softmax'))(conv9)
model = Model(inputs, conv10)
model.outputWidth = outputWidth
model.outputHeight = outputHeight
return model
训练请看: https://github.com/BBuf/Keras-Semantic-Segmentation
本文总阅读量次