MICCAI 2023:Continual Learning 的腹部多器官和肿瘤分割¶
目录¶
- Continual Learning 介绍
- 概述
- 具体实现(Methodology)
- 问题定义
- 伪标签设置
- 模型细节
- 计算复杂度
- 实验
- 总结
- 参考
Continual Learning 介绍¶
在介绍这篇文章的方法之前,我们先来简单引入一下 Continual Learning 的概念。

在各种各样的任务里,基于学习的方法都能够获得很好的表现,但是有一个前提条件是,训练集的分布和测试集的分布要一致或者相似。也就是说,当环境发生变化时,系统并不能很好地适应。而对于真正的智能系统来说,这种适应是必不可少的。比如,当我们学会分辨现实中的狗和猫以后,我们也能轻松地分辨动画片中的狗和猫。

其中一种关于智能系统适应的研究方向是 Continual Learning,持续学习的设定是随着训练数据流的流入,模型不断地增量学习(意味着我们不能一次性地已知所有的训练数据和任务,而是每流入一份数据和对应任务就开展一次学习),最终完成对所有任务的训练。尽管深度神经网络模型在各种各样的任务中都表现得非常好,但当模型通过梯度下降算法来增量地学习时,往往会经受非常严重的干扰以及遗忘之前学习过的知识。换言之,当模型在新的任务中进行参数更新时,由于受到参数更新的干扰,模型会忘记如何解决旧任务。而持续学习就是主要研究怎样去解决这种干扰和遗忘问题。Transfer Learning 与持续学习不同的是,迁移学习研究的是如何把旧任务中所学习的知识迁移到新任务中,主要关注的是新任务中的表现。而持续学习既要关注新任务上的的表现,又要关注在旧任务上的表现。
概述¶
在医学领域,将模型动态扩展到新类别对于多器官和肿瘤分割至关重要,其中的关键障碍在于减轻“遗忘”。一种典型的策略涉及保留一些先前的数据。例如,引入了一个内存模块,用于存储不同器官类别的原型表示。然而,依赖于数据和注释的方法可能会面临实际约束,因为隐私问题可能会使获取先前数据和注释变得困难。还有一些研究主要集中在架构扩展方面,通过冻结编码器和解码器以及在学习新类别时添加额外的解码器来解决遗忘问题。虽然这些策略已经缓解了遗忘问题,但它们导致了模型参数的巨大内存成本。因此,我们确定了在设计多器官和肿瘤分割框架时必须解决的两个主要开放问题。
- 问题1:是否可以在不需要先前的数据和注释的情况下减轻遗忘问题?
- 问题2:是否可以设计一种新的模型架构,能够在不同的持续学习步骤之间共享更多的参数?
为了解决上述问题,本文提出了一种新颖的持续多器官和肿瘤分割方法,该方法通过很少的内存和计算开销克服了遗忘问题。首先,受到持续学习中的知识蒸馏方法的启发,提出在新到达的数据上为旧类别生成软伪标签。这使得能够在不保存旧数据的情况下回忆旧知识(这类方法已经有了)。通过这种简单的策略,能够保持对旧类别的合理性能。其次,为每个类别提出了基于图像的分割头部,位于共享编码器和解码器之上。这些头部允许使用单个骨干,并轻松扩展到新类别,同时带来较少的计算成本。采用了由 CLIP 生成的文本嵌入,CLIP 是一个大规模的图像-文本联合训练模型,能够将高级视觉语义编码到文本嵌入中。这些信息将有助于使用事先已知的类别名称来训练新的分割类别(可扩展性强)。
在这之前我们在 GiantPandaCV 公众号上已经介绍过一个工作,文章题为【ICCV 2023:CLIP 驱动的器官分割和肿瘤检测通用模型】,可以搜索到。这两篇文章的架构基本一致,后文我们会介绍他们的微弱区别。
具体实现(Methodology)¶
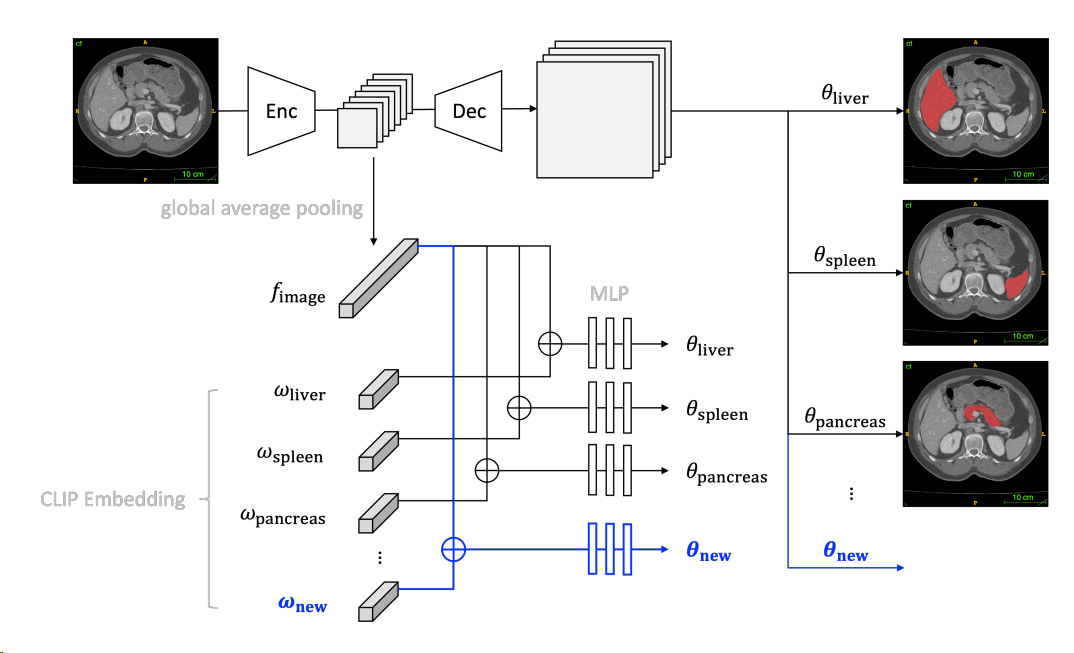
下图是整体框架的实现,一个编码器(Enc)处理输入图像以提取其特征,然后通过全局平均池化层将其减少为特征向量(fimage)。将此特征向量与使用预训练的 CLIP 模型计算的 CLIP embedding(ωclass)连接在一起。通过一系列多层感知器(MLP)层,得到卷积核(θclass)的特定类别参数。这些卷积核应用于解码器(Dec)特征时,产生了相应类别的掩码。
问题定义¶
将连续分割的形式化表示如下:给定一系列部分注释的数据集 {D1,D2,...,Dn},每个数据集都包含器官类别 {C1,C2,...,Cn},依次使用一个数据集来逐个学习单一的多器官分割模型。在训练第 i 个数据集 Dt 时,之前的数据集 {D1,...,Dt−1} 不可用。模型需要预测所有已见数据集 {D1,...,Dt} 的累积器官标签: $$ \begin{aligned} \hat{Y}j & =\underset{c \in \mathcal{C}_t}{\operatorname{argmax}} P\left(Y_j=c \mid X\right) \ \mathcal{C}_t & =\cup{\tau \leq t} C_\tau \end{aligned} $$ 其中 j 是体素索引,X 是来自数据集 Dt 的图像,P 是模型学习的概率函数,\hat{Y}_{j}是输出的分割掩码。
伪标签设置¶
我们在概述中提到的一个需要解决的问题是模型遗忘问题,这篇工作发现使用伪标签可以在很大程度上缓解这个问题并保留现有的知识。具体而言,利用了前一次学习步骤 t−1 的输出预测,表示为 \hat{Y}_{t-1},其中包括旧类别 Ct−1,作为当前学习步骤的旧类别的伪标签。对于新类别,仍然使用地面实际标签。形式上,当前学习步骤 t 中类别 c 的标签 \tilde{L}_t^c 可以表示为: $$ \tilde{L}t^c= \begin{cases}L_t^c & \text { if } c \in \mathcal{C}_t-\mathcal{C}{t-1} \ \hat{Y}{t-1}^c & \text { if } c \in \mathcal{C}{t-1}\end{cases} $$ 其中 L_t^c 表示从数据集 Dt 中获得的类别 c 在步骤 t 中的标签。通过采用这种方法,防止模型在学习新类别的同时遗忘先前学到的信息。这样提出的模型只使用了旧类别的伪标签进行训练,没有其他蒸馏或正则化。
也就是说,我们在训练网络时使用的数据不再只是当前类别,而是包括之前遇到的类别(只不过是伪标签)。这种解决遗忘的方式很简单,但是不可避免的是要么需要更大的显存,要么需要训练更长时间。考虑到推理速度没有影响,是可以接受的。
模型细节¶
Backbone:对于 continual learning,理想情况下,模型应该能够学习一个足够通用的表示,以便轻松适应新的类别。选择使用 Swin UNETR 作为主干模型,因为它在自监督预训练和在各种医学图像分割任务中的迁移性能上表现出色。Swin UNETR 的编码器采用 Swin Transformer,解码器则包括多个反卷积层。
图像感知器官特异性 head:我们知道,标准的 Swin UNETR 具有一个 Softmax 层作为输出层,用于预测每个类别的概率。将输出层替换为多个图像感知器官特定 head。首先,在最后一个编码器特征上使用全局平均池化(GAP)层,以获取当前图像 X 的全局特征 f。然后,对于每个器官类别 k,学习一个 MLP 模块,将全局图像特征映射到一组参数 θk: $$ \theta_k=\operatorname{MLP}_k(\operatorname{GAP}(E(X))) $$ 其中 E(X) 表示图像 X 的编码器特征。针对器官类别 k 的输出头部是一系列卷积层,使用参数 θk 作为卷积核参数。这些卷积层应用于解码器特征,输出器官类别 k 的分割预测: $$ P\left(Y_j^k=1 \mid X, \theta_k\right)=\sigma\left(\operatorname{Conv}\left(D(E(X)) ; \theta_k\right)\right) $$ 其中 E 是编码器,D 是解码器,σ 是 Sigmoid 非线性层,P\left(Y_j^k=1\right) 表示体素 j 属于器官类别 k 的预测概率。每个类别的预测通过二元交叉熵损失进行优化。独立的头部允许对新引入和先前学习的类别进行独立的概率预测,因此在 continual learning 过程中最小化新类别对旧类别的影响。此外,这种设计还允许多标签预测,用于像一个器官上的像素属于多个类别的情况(Sigmoid)。
CLIP 驱动 head 参数生成:这一部分内容我们在【ICCV 2023:CLIP 驱动的器官分割和肿瘤检测通用模型】中介绍过了,读者可以先阅读这个前置文章。最后,生成参数 θk 可以修改为: $$ \theta_k=\operatorname{MLP}_k\left(\left[\operatorname{GAP}(E(X)), \omega_k\right]\right), $$ 与前置文章的不同之处:为了实现 continual learning 的目的,在 MLP 模块的原始设计上进行了改进。 前置文章使用单一的 MLP 来管理多个类别,这篇工作为每个类别分配了一个独立的 MLP,这个设计可以显著减轻不同类别之间的干扰。
计算复杂度¶
与基线模型 Swin UNETR 进行了比较。这篇工作的模型 FLOPs 略高于 Swin UNETR 的 FLOPs,分别为 661.6 GFLOPs 和 659.4 GFLOPs。这是因为使用了具有少量通道的轻量级输出卷积头。对比之前用于医学持续语义分割的架构,该架构使用了一个经过预训练并冻结的编码器,以及在每个学习步骤中逐渐添加的解码器。然而,使用这种架构进行后续的持续学习步骤引入了巨大的计算复杂性。例如,Swin UNETR 的解码器单独就有 466.08 GFLOPs,这意味着每个新的学习步骤都会增加 466.08 GFLOPs。相比之下,这篇工作的模型只需要为新任务的新类别添加一些图像感知的器官特定头部,每个头部只消耗 0.12 GFLOPs。因此,在分割的 continual learning 中几乎保持了计算复杂性基本不变,而之前的架构的计算复杂性随着步骤数的增加而线性增加。此外,与比需要进行特征蒸馏(会使 FLOPs 翻倍)的方法比较,也有很大的优势,
实验¶
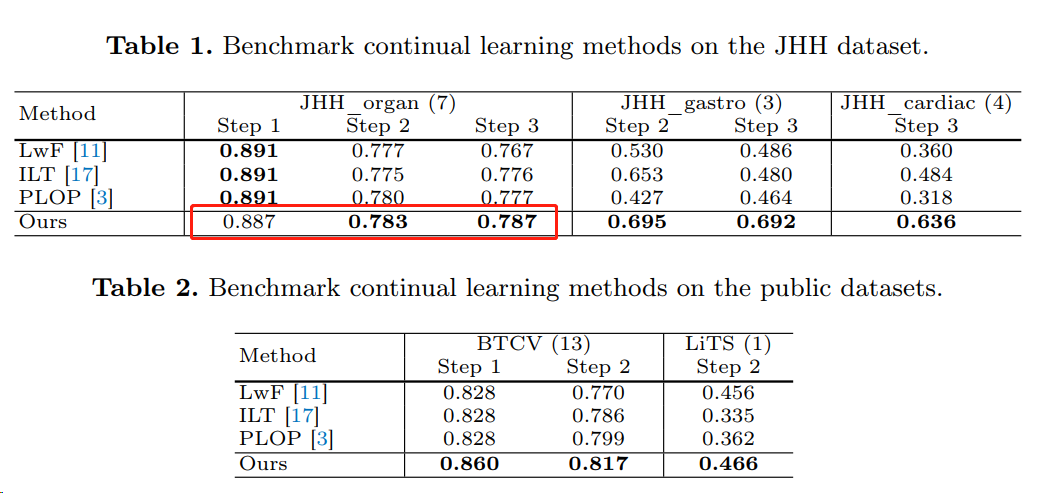
从下表 1 可以发现,这篇工作的方法虽然在 step 1 的性能轻微低,但是在后面的 step 性能有所提升。说明缓解了 continual learning 的遗忘问题。
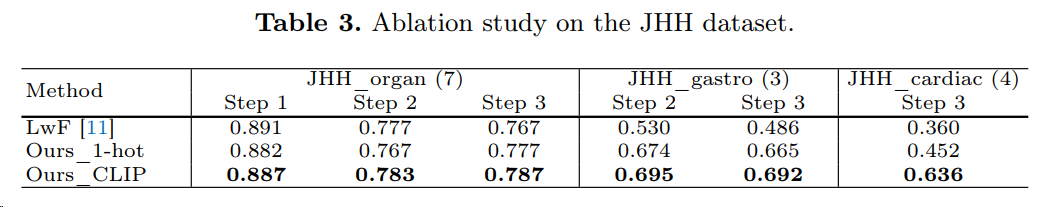
下表说明了使用 CLIP 编码 label,要好于 one-hot 编码,这在前置文章中也有实验。
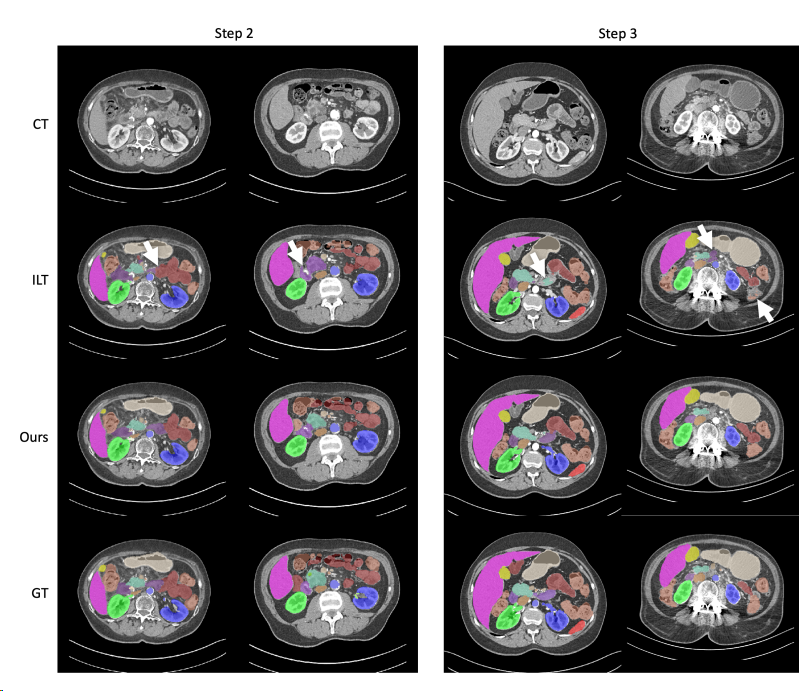
可视化结果如下图:
总结¶
这篇文章介绍了一种针对医学图像分割的 continual learning 方法,主要解决了在添加新类别时避免遗忘旧知识的问题。作者提出了使用伪标签和图像感知头部(基于 CLIP)的创新方法,有效降低了计算复杂性,并在实验中取得了良好的分割性能,为医学图像分割领域的 continual learning 提供了有前景的解决方案。
参考¶
本文总阅读量次